La IA revela una biología previamente desconocida: es posible que no sepamos la mitad de lo que hay en nuestras células

Los investigadores de UC San Diego presentan la célula integrada multiescala (MuSIC), una técnica que combina microscopía, bioquímica e inteligencia artificial, revelando componentes celulares previamente desconocidos que pueden proporcionar nuevas pistas sobre el desarrollo y la enfermedad humanos. (Representación conceptual del artista) Crédito: UC San Diego Health Sciences
La técnica basada en inteligencia artificial revela componentes celulares previamente desconocidos que pueden proporcionar nuevas pistas sobre el desarrollo y las enfermedades humanas.
La mayoría de las enfermedades humanas se remontan a partes defectuosas de una célula: un tumor puede crecer porque un gen no se ha traducido con precisión en una proteína específica, o surge una enfermedad metabólica porque las mitocondrias no se activan correctamente, por ejemplo. Pero para comprender qué partes de una célula pueden fallar en una enfermedad, los científicos primero deben tener una lista completa de las partes.
Al combinar microscopía, técnicas bioquímicas e inteligencia artificial, los investigadores de la Facultad de Medicina de la Universidad de California en San Diego y sus colegas han logrado lo que creen que podría ser un avance significativo en la comprensión de las células humanas.
La técnica, conocida como Multi-Scale Integrated Cell (MuSIC), se describe el 24 de noviembre de 2021, en Naturaleza.
“Si imagina una célula, probablemente imagina el diagrama de colores en su libro de texto de biología celular, con mitocondrias, retículo endoplásmico y núcleo. ¿Pero es esa toda la historia? Definitivamente no ”, dijo Trey Ideker, PhD, profesor de la Facultad de Medicina de UC San Diego y del Moores Cancer Center. “Los científicos se dieron cuenta hace mucho tiempo de que hay más cosas que no sabemos de las que sabemos, pero ahora finalmente tenemos una manera de mirar más a fondo”.
Ideker realizó el estudio con Emma Lundberg, PhD, del KTH Royal Institute of Technology en Estocolmo, Suecia y la Universidad de Stanford.
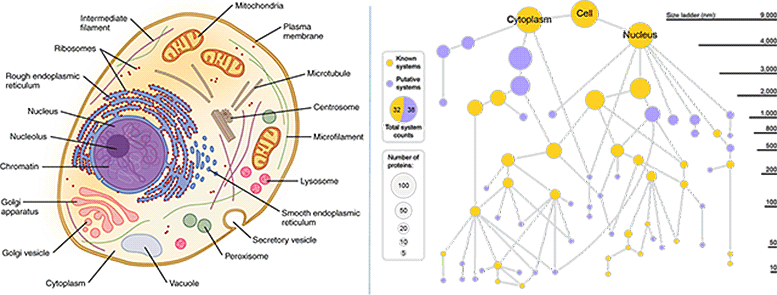
Izquierda: los diagramas de celda clásicos de los libros de texto implican que todas las partes son claramente visibles y definidas. (Crédito: OpenStax / Wikimedia). Derecha: Un nuevo mapa de células generado por la técnica MuSIC revela muchos componentes nuevos. Los nodos dorados representan componentes conocidos de la celda, los nodos violetas representan componentes nuevos. El tamaño del nodo refleja el número de proteínas distintas en ese componente. Crédito: UC San Diego Health Sciences
En el estudio piloto, MuSIC reveló aproximadamente 70 componentes contenidos en una línea celular de riñón humano, la mitad de los cuales nunca se habían visto antes. En un ejemplo, los investigadores localizaron un grupo de proteínas que forman una estructura desconocida. Trabajando con el colega de UC San Diego, Gene Yeo, PhD, finalmente determinaron que la estructura era un nuevo complejo de proteínas de unión. ARN. Es probable que el complejo esté involucrado en el empalme, un evento celular importante que permite la traducción de genes en proteínas y ayuda a determinar qué genes se activan en qué momento.
El interior de las células, y las muchas proteínas que se encuentran allí, generalmente se estudian utilizando una de dos técnicas: imágenes de microscopio o asociación biofísica. Con la imagen, los investigadores agregan marcas fluorescentes de varios colores a las proteínas de interés y siguen sus movimientos y asociaciones en el campo de visión del microscopio. Para examinar las asociaciones biofísicas, los investigadores pueden usar un anticuerpo específico de una proteína para sacarla de la célula y ver qué más está ligada a ella.
El equipo ha estado interesado en mapear el funcionamiento interno de las células durante muchos años. Lo que es diferente acerca de MuSIC es el uso del aprendizaje profundo para mapear la célula directamente a partir de imágenes de microscopía celular.
“La combinación de estas tecnologías es única y poderosa porque es la primera vez que se reúnen medidas a escalas muy diferentes”, dijo el primer autor del estudio, Yue Qin, estudiante graduado en Bioinformática y Biología de Sistemas en el laboratorio de Ideker.
Los microscopios permiten a los científicos ver hasta el nivel de una sola micra, el tamaño de algunos orgánulos como las mitocondrias. Los elementos más pequeños, como las proteínas individuales y los complejos de proteínas, no se pueden ver al microscopio. Las técnicas bioquímicas, que comienzan con una sola proteína, permiten a los científicos descender a la nanoescala. (Un nanómetro es una mil millonésima parte de un metro, o 1000 micrones).
“Pero, ¿cómo se llena ese espacio entre la escala nanométrica y la escala micrónica? Este ha sido un gran obstáculo en las ciencias de la vida ”, dijo Ideker, quien también es fundador de UC Cancer Cell Map Initiative y el Centro de Biología Computacional y Bioinformática en UC San Diego. “Resulta que puedes hacer esto con inteligencia artificial: ver datos de múltiples fuentes y pedirle al sistema que los monte en un modelo celular”.
El equipo entrenó la plataforma de inteligencia artificial MuSIC para examinar todos los datos y construir un modelo de la célula. El sistema aún no asigna el contenido de las celdas a ubicaciones específicas, como un diagrama de libro de texto, en parte porque sus ubicaciones no son necesariamente fijas. En cambio, las ubicaciones de los componentes son fluidas y cambian según el tipo de celda y la situación.
Ideker señaló que se trataba de un estudio piloto para probar MuSIC. Observaron solo 661 proteínas y un tipo de célula.
“El siguiente paso claro es atravesar toda la célula humana”, dijo Ideker, “y luego pasar a diferentes tipos de células, personas y especies. Con el tiempo, es posible que podamos comprender mejor la base molecular de muchas enfermedades comparando las diferencias entre las células sanas y las enfermas. “
Referencia: “Un mapa multiescala de la estructura celular que fusiona imágenes e interacciones de proteínas” por Yue Qin, Edward L. Huttlin, Casper F. Winsnes, Maya L. Gosztyla, Ludivine Wacheul, Marcus R. Kelly, Steven M. Blue, Fan Zheng, Michael Chen, Leah V. Schaffer, Katherine Licon, Anna Bäckström, Laura Pontano Vaites, John J. Lee, Wei Ouyang, Sophie N. Liu, Tian Zhang, Erica Silva, Jisoo Park, Adriana Pitea, Jason F. Kreisberg , Steven P. Gygi, Jianzhu Ma, J. Wade Harper, Gene W.Yeo, Denis LJ Lafontaine, Emma Lundberg y Trey Ideker, 24 de noviembre de 2021, Naturaleza.
DOI: 10.1038 / s41586-021-04115-9
Los coautores incluyen: Maya L. Gosztyla, Marcus R. Kelly, Steven M. Blue, Fan Zheng, Michael Chen, Leah V. Schaffer, Katherine Licon, John J. Lee, Sophie N. Liu, Erica Silva, Jisoo Park, Adriana Pitea, Jason F. Kreisberg, UC San Diego; Edward L. Huttlin, Laura Pontano Vaites, Tian Zhang, Steven P. Gygi, J. Wade Harper, Facultad de Medicina de Harvard; Casper F. Winsnes, Anna Bäckström, Wei Ouyang, Instituto Real de Tecnología de KTH; Ludivine Wacheul, Denis LJ Lafontaine, Université Libre de Bruxelles; y Jianzhu Ma de la Universidad de Pekín.
Los fondos para esta investigación fue, en parte, de los Institutos Nacionales de la Salud (Subvenciones U54CA209891, U01MH115747, F99CA264422, P41GM103504, R01HG009979, U24HG006673, U41HG009889, R01HL137223, R01HG004659, R01HCA, R01G00H00H5659, R01G01, Alice R01G01G01H0659, R01G01 R01HG00CA, R01HG00CA y R01HG004659 ). Fundación Wallenberg (subvención 2016.0204), Consejo de Investigación de Suecia (subvención 2017-05327), Belga Fonds de la Recherche Scientifique, Université Libre de Bruxelles, Programa Conjunto Europeo sobre Enfermedades Raras, Région Wallonne, Internationale Brachet Stiftung y Epitran COST action (subvención CA16120)
Divulgaciones: Trey Ideker es cofundador del Scientific Advisory Board y es un accionista de capital en Data4Cure, Inc. Ideker también está en el Scientific Advisory Board, es un accionista de capital y recibe fondos de investigación patrocinados de Ideaya BioSciences, Inc. Gene Yeo es cofundador, miembro del Consejo de Administración, Consejo Asesor Científico, accionista y consultor remunerado de Locanabio y Eclipse BioInnovations. Yeo también es profesor invitado en la Universidad Nacional de Singapur. Los términos de estos acuerdos han sido revisados y aprobados por la Universidad de California en San Diego de acuerdo con sus políticas de conflicto de intereses. Emma Lundberg es miembro de los Consejos Asesores Científicos y tiene una participación accionaria en Cartography Biosciences, Nautilus Biotechnology e Interline Therapeutics. J. Wade Harper es cofundador del Consejo Asesor Científico y participa en el capital social de Caraway Therapeutics. Harper es también asesor científico fundador de Interline Therapeutics.

“Creador malvado. Estudiante. Jugador apasionado. Nerd incondicional de las redes sociales. Adicto a la música”.




